什么?细胞还要身份证?
我们知道,细胞是构成我们人体的基本单位。可是你知道吗?相比于构成细胞的原子、分子,我们恰恰对细胞本身知之甚少。人体里面到底有多少种细胞?它们是如何分布的、如何相互作用的?一直以来,这些问题的答案与人体的很多生命作用息息相关,引发着人们的好奇,但探索起来也困难重重,朦朦胧胧。
为什么对细胞的了解这么有用呢?
我们的身体里面三十万亿个细胞,而这些细胞的种类和分布,它们之间怎么对话、怎么协同完成一些生命过程,我们的了解非常有限。而这些个信息呢,直接关系着人体的奥秘。比方说吧,每个人都会生病,对于很多疾病来说,这个发病的过程就是相应细胞变化的过程,往往在这个过程里,某些细胞的功能、形态、特性都会发生改变。甚至对于像癌症一类的疑难杂症,肿瘤细胞附近很多种细胞都会发生变化,肿瘤细胞和体内正常的细胞(如免疫细胞)还会有很多相互作用。如果我们对这些细胞的种类、分布、分化、相互作用有了深入的了解,我们就可以揭示更多的生命奥秘。
科学家盯上RNA
到底怎么给细胞进行细致的分类和描述呢?科学家想到了RNA。
分子生物学中,有个“中心法则”,说的是遗传物质从载体——DNA传递给RNA, RNA再翻译成蛋白质,即完成遗传信息的转录和翻译的过程。对于同一个人来说,不同种类的细胞DNA是相同的,而细胞之所以有各种变化万千的形态和功能,是因为不同细胞里RNA表达和修饰不一样。这些各种各样的RNA是遗传物质DNA转录的产物,人们称其为转录组。而通过对细胞转录组做一个定量分析来测绘细胞种类、甚至描述它们空间分布和相互作用的方法,就叫做转录组的成像。
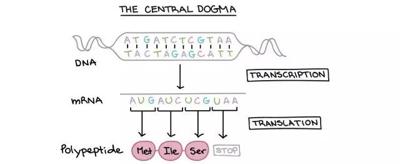
但是呢,这种美好的想法只是看上去很美,实现起来却困难重重。因为RNA的种类太多了!
在一个细胞里,基因有上万个,而RNA的数目比基因的数目还多若干倍。传统方法怎么做呢?1998年,阿尔伯特爱因斯坦医学院的RobertSinger研究组发明了单分子荧光原位杂交方法(smFISH),这种方法是用荧光标记各种DNA或者RNA,再将这些带了荧光的DNA或者RNA作为探针去探测细胞中的某一特定序列的RNA,以此来检测细胞中某一特定RNA的数量、定义它们的位置。
实际情况中,通过结合不同荧光的探针,这种方法可以同时检测30种不同的RNA分子。但是聪明的你一定看出问题来了——一个细胞中会有好几万种RNA,如果科学家们想同时检测更多的RNA分子,比如上万种不同的RNA该如何呢?难道我们能使用6万种不同颜色的探针去检测嘛?显然绝对做不到。
RNA玩转二进制 编码
颜色的标记办法行不通,科学家开始想其它主意。2005年,庄小威研究组从电子通信领域获得灵感,借助二进制编码的理念,发明了全新的方法去检测细胞中的RNA水平,可以同时检测达六万种RNA,这种方法叫MERFISH(mutiplexed error-robust fluorescence in situ hybridization)。
我们之前已经说过,单分子荧光原位杂交可以用来看某一种RNA。所以,一个RNA可以用二进位标码来标记——被探针结合的细胞会被点亮(标记为1),其余没有结合的不会亮(标记为0)。这就是第一回合的采集,形成第一个图像;接着,采集第二个image,我们读第二个RNA的二进制标码。每一次的采集会有不同的细胞会被点亮,而每一次反馈的读数都由二进制编码来标记(0或者1)。这样重复下去,这组探针结合到细胞中的各个目标RNA之后会反馈出一组读数。读到N位以后,会得到这样一个图像,把每一轮的标记(0或1)串在一起就成了每一种RNA的条码,每一种RNA都有自己特定的条码。把这些点亮的点全部放到一起,就能得到图一左下的图,其中每一种颜色的点代表一种条码,即一种RNA(图一)。
MERFISH是一个高度多重化的smFISH成像方法。我们来做一个简单的数学题,所以对于一个特定的细胞来说,两次采集数据后所反馈的读数有4种可能性,分别是00,01,10,11,依次类推,当N次采集数据后所反馈的读数是2的N次方。如果N做到16,就可以轻松区分6万种可能性,人体的转录组轻松破解。真的这么乐观吗?
情况远没有这么理想,因为单分子荧光原位杂交这种方法再精确,也是有误码率的。即使这个误码率只有5%,测到第二回合,误码率就将近10%,N达到16的时候,误码率也会大大放大。为此,该实验小组信息时代又借鉴了现代的信息学理念,发展了一种叫容错纠错(error-robust)编码的策略,他们只使用与其他代码至少相差两个或以上比特(二进制单位)的代码,这种方法虽然限制了可检测的不同RNA的多样性(丰度),但却大大提高了准确率。这种想法在转录组成像技术上是前所未有的。
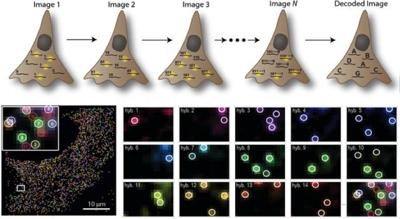
图1 MERFISH方法
http://www.hhmi.org/news/new-method-allows-precise-measurement-transcriptome-single-cells
MERFISH galle ry
我们一起来透过MERFISH方法来看一看生命细胞吧。
首先,来一个人肺的成纤维细胞系(图二)。科学家用16比特MHD4代码[1]的MERFISH方法同时检测单个细胞中的140种RNA。
A图是每一次用探针杂交样品后的图像以及光漂白(photobleaching)后的图像。阐释这种方法可以有效地去除相邻杂交成像之间的荧光信号,也就是MERFISH方法的必要条件。
B图是经过16回合的杂交成像后(每一种代码表示一种颜色即一种RNA,也就是每一个特意代码的RNA上了伪色,便于分析数据),所得到的所有分子的信息。其中7号红色圈指无法辨认的信号,也就是在16进MHD4代码中没有符合的代码,即便用error correction排除潜在错误信号后仍然存在的错误。所以说MERFISH的方法中所用到的错误排除法并不是100%的,即便排除错误仍然会存在一些无法辨认的信号。
C图是取了B图的一部分,用来展示每一次杂交成像后的潜在的RNA分子。

图2 MERFISH方法同时检测单个细胞中的140种RNA
如果我们用MHD2呢?牺牲了准确度,但是可以同时检测的RNA就多了。图三中是用14进MHD2代码的MERFISH同时在单个细胞中检测1001种RNA。和图二一样,每一种伪色代表的是一种RNA分子,在14回合的杂交成像后所的到的信息,同样,红色圈是模棱两可的信息。

图3 用14进MHD2代码的MERFISH同时在单个细胞中检测1001种RNA
图片来源:
http://science.sciencemag.org/content/348/6233/aaa6090/tab-figures-data[2]
MERFISH除了可以成像单个细胞中的RNA,我们还可以利用它检测染色体(DNA)的空间定位。什么叫染色体的空间定位呢?在真核细胞中,DNA被包装成称作为染色质的复杂大分子结构,而染色质的空间组织严重影响了基因组功能。
近期一些染色体研究发现,单条染色体被分割成长度在数十千碱基(kb)到数兆碱基(Mb)之间的接触结构域或拓扑相关结构域(topologically associating domains,TADs)。
TADs作为非常长的DNA片段,包含着一个或多个基因及其调控元件。TADs是染色质组织的一个保守特征,它的一个重要功能就是形成基因调控的独立区域,同时将它们与邻近区域隔离开来(图四)。如果染色质重排影响TADs的组织形式,那么许多基因的表达都会发生改变,甚至引发多种人类疾病。

图4 TADs示意图
图片来源:Wikipedia
那么研究人员如何利用MERFISH绘制基因组区域的空间位置呢?我们可以想象以下,要想从外太空定位中国的位置,首先需要定位地球,再从地球上找到中国的位置。
科研人员再次利用人肺的成纤维细胞系(IMR90细胞系),尝试用MERFISH方法检测单个成纤维细胞中21号染色体中心100kb区域中的34个TADs的空间定位。
由于人的细胞中有23对染色体(除减数分裂后的细胞以外),所以如果想检测其中21号染色体,我们首先需要定位它的位置。好在通过全基因组测序,我们已经掌握了21号染色体中34个TADs的序列,通过这些信息可以制作专门识别21号染色体上TADs的带荧光的探针,我们可以把这些探针叫做初级探针。
好了,现在我们用初级探针定位到了“地球”的位置,那么下面我们该如何定位“中国”呢?
初级探针实际上除了有与DNA结合的互补区,还有延伸段用来与二级探针结合。二级探针上也偶联了荧光分子,我们可以使用两种不同的荧光,这样只需要17回杂交就可以得到34个TADs的位置。
图五中,A图是这种方法的成像策略;B图是在结合一级探针后的成像,细胞中常染色体是成对的,所以有两个光斑,也就是两个21号染色体;C图是B图的黄色框中的部分,在17轮杂交成像中每一轮成像后的图像;D和E图是21号染色体上34个TADs的二维和三维分布图。
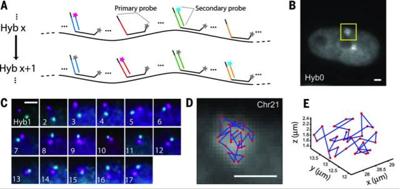
图5 21号染色体及其上面34个TADs的空间分布
虽然目前这种MERFISH方法不是绝对完美,但是它可以通过给单个细胞一个特定的“身份证号”,来帮助我们识别不同种类的细胞。
在人类跟可怕的疾病——比如癌症斗争的过程中,认识和了解不同种类癌细胞的特性是一个巨大的挑战,只有了解了不同种类细胞的发生、生长、扩散、转移以及药理机制,才能有针对性的给病人最好的治疗。
这是一个宏大的课题,庄小威教授曾做过一个比喻:“人体内到底有多少种不同的细胞,这些细胞在空间上是如何分布和相互作用的,对于我们想要掌握人体生命奥秘的人来说,是必须要去了解的图像。如果把人体比作汽车,那么所有的零件一一拆开,你无法知道他们是怎么运转的。只有将他们在空间上组装起来,你才能知道它为什么能飞快奔跑。”
参考文献及说明
[1]MHD全称是modified Hammington distance,MHD4是汉明距离等于四。因为用二进制方法,杂交N次后得到的RNA丰度应该是2N-1,但如果这样的话N次杂交结果中有一次出错就可能代表另一种RNA了,这样错误率很高,所以他们把汉明距离改成4,也就是一窜二进制代码里面至少有4位不一样才算是不同的RNA,这样虽然降低了丰度,但错误率就减少了。
[2]Chen, K.H., et al., RNA imaging. Spatially resolved, highly multiplexed RNA profiling in single cells. Science, 2015. 348(6233): p. aaa6090.
[3] Wang, S., et al., Spatial organization of chromatin domains and compartments in single chromosomes. Science, 2016. 353(6299): p. 598-602.



