
图1 肌动蛋白骨架
图片来源:2012, Nature Methods
生命的尺度
我们知道,人体是由许许多多的细胞构成的,细胞,则是由分子构成,而分子是由原子构成的。在不同的尺度上,原子、分子、细胞遵循一定的排列规则,形成一定的结构,完成特定的功能。如果我们可以知道这些分子和细胞是怎么排布的,甚至看到它们的工作过程,那么对于生命的了解一定会加深许多。
但是,想要看见这个过程并不容易。
说起观测生物的微观结构,我们首先会想到三个字——显微镜。的确,自从17世纪人们发明显微镜,微小的内部结构呈现在人们眼前以来,生物学和医学才得有了较大的进步。比如说,过去一场外科手术,患者往往并不是死于疾病本身,而是死于莫名其妙的感染,在显微镜的帮助下,人们见识了导致感染的各种微生物,今天,无菌术成为保障我们医疗安全的常见手段。
可惜,在想无限提高放大倍数的过程中,我们沮丧地发现,显微镜的放大倍率是有极限的。这种极限来自于光的衍射效应对于分辨率的影响。
我们中学就学过,光是一种波,波都有衍射效应。这意味着,当你把光聚焦到一点上时,哪怕用最理想的聚焦手段,这个光斑也有一定大小。这个尺寸最小能到多小呢?德国科学家E.Abbe在1873年告诉我们,大约在200nm的样子。200nm虽然很小,但是与通常不超过10nm的分子相比,还是太大了。当达到显微镜分辨率极限的时候,即使放大倍率进一步提高,得到的也只能是一个轮廓稍大但细节不清的图像。为什么说细节不清呢?我们可以想象一下,假如你想要绣花,你手中拿着细细的绣花针,当然可以上下翻飞游刃有余,可你要是拿着筷子那么粗的大棍子(200nm的光斑),还能绣出这么细致的图案(10nm的分子)吗?
人们希望找到一些方法,能突破这个光学上的极限。
突破极限
为了突破衍射极限,人们从两个角度思考这个问题。一是光的角度,二是样品的角度,并由此衍生出很多方法。
从光的角度,人们发明了很多技术,比如近场扫描光学显微镜(NSOM,near-field scanning optical microscopy)、多光子荧光(multiphoton fluorescence)、受激发射损耗荧光显微术(STED,stimulated emission depletion)、饱和光照明显微镜(SSIM,saturated structured-illumination microscopy)等。他们的原理各有千秋,优劣也不尽相同。
比如,NSOM探测的是样品表面很近的非辐射场(即样品表面附近的光场)的信息,所以虽然突破了衍射极限,但是成像深度(imaging depth)很浅。
又比如,大名鼎鼎的STED,设计原理也非常巧妙,主要是利用一束光激发样品分子,使电子跃迁到一个高轨道,再用另一束光将光斑外围的电子打到一个低轨道上,光斑中心的电子让它自然回到基态,由于这两个过程放出的光子波长不同,我们就能让探测器只看到光斑中心的信息啦。

图2 Stefan W. Hell,STED的发明者,荣获2014年诺贝尔化学奖
图片来源:https://www.nobelprize.org/nobel_prizes/chemistry/laureates/2014/press.html
不过呢,multiphoton microscopy、STED和SSIM都是基于非线性光学效应(nonlinear optical effects)的,所以需要很强的脉冲激光,而这会导致样品的损伤。而这种光诱导的损伤导致生物样品中的蛋白质构象不同于自然状态(native state),因此通过显微镜看到的可能也是“变了形”的状态。
从样品的角度上也有一些优化的方法,比如单纳米精度荧光成像(FIONA,fluorescence imaging with one-nanometer accuracy)方法,就是是通过收集足够多的光子来判断单分子发光体(emitter)的位置,并且它的准确率非常高,同时它可以在室温中进行实验。但是FIONA中探测到的位置信息无法直接转换成图像分辨率,如果两个发光体(emitter)的距离靠的很近时依然无法得到准确的位置信息。

图3 William E. Moerner,第一个能够探测单个荧光分子的人,荣获2014年诺贝尔化学奖
图片来源:https://www.nobelprize.org/nobel_prizes/chemistry/laureates/2014/press.html
看来,单分子探测技术(single-molecule detection)还需要进一步提高分辨率。怎么做呢?人们想到了这样一个思路:
所谓的衍射极限是怎么影响分辨率的呢?就是说当你想盯住一个点看的时候,光斑却有一定大小,所以成像也有一定大小,当两个及以上的点离得很近时,像和像就会有一部分重叠在一起,看起来是一个大光斑而不是几个小光斑。那么科学家就想了,这没关系,虽然成像有宽度,但是像的中心点还是很好找的。如果能对距离相近的分子成像在时间上拉开距离,不让它时间上重叠,这个位置先亮、那个位置后亮,不就能区分谁是谁了吗?
具体怎么做呢?科学家在不同的分子上安了“开关(photoswitching property)”,每次成像,开一小部分分子,让它们发光,然后关掉,再开一小部分分子让它们发光,这样经过很多很多次,每个分子位置都可以被精确的记录下来,将所有的成像叠加,一个清晰的高分辨图像就呈现了。
2006年,三个小组(其中之一是咱们中国科大的校友庄小威教授领导的研究小组)都独立发表了这样的方法[1,2,3],人们将这样的方法叫做Photo-activated localization microscopy(PALM) 或者stochastic optical reconstruction microscopy(STORM)。

图4 Eric Betzig,发明PALM荣获2014年诺贝尔化学奖
图片来源:https://www.nobelprize.org/nobel_prizes/chemistry/laureates/2014/press.html
后来,庄小威教授对这种方法进行了进一步的改进和应用,比如做到了三个维度同时的超分辨率显现,这样就可以看到生物分子的三维结构;又比如进一步提高了该技术的成像速率,快速的时间分辨率为观察活体的动态成像提供了帮助。

STORM Gallery
我们一起来感受这项技术能带给我们的美妙和震撼吧。
1.精子鞭毛中的CatSper [2014, Cell]

通过超高分辨率显微镜,科学家们发现了精子鞭毛中组织信号蛋白的四个线性钙离子结构域由CatSper通路所控制,而这种独特的结构使得在时间上和空间上集中于酪氨酸的磷酸化,从而使精子获得受精的能力。而只有具备完整的CatSper结构域的精子才可以成功地进行迁移。
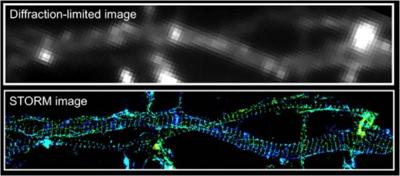
2.轴突的周期性细胞支架 [2013, Science]
比较普通显微镜(上图)和3D STORM(下图)拍摄神经元中轴突的肌动蛋白。STORM图像阐释了一个轴突中由肌动蛋白-血影蛋白为单位的周期性的膜骨架。
3.突触的骨架 [2015, Cell]

STORM动画中展示的是视网膜神经节细胞与其抑制突触输入区。突触由突触前蛋白(红色)和gephrin(绿色)标记。神经元可以表达绿色荧光蛋白(GFP),并且使用带光控染料(photoswitchable dye)的抗-GFP抗体标记。
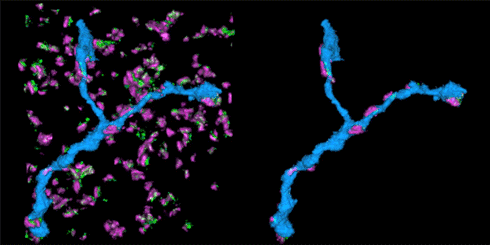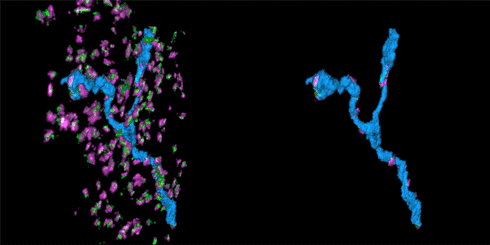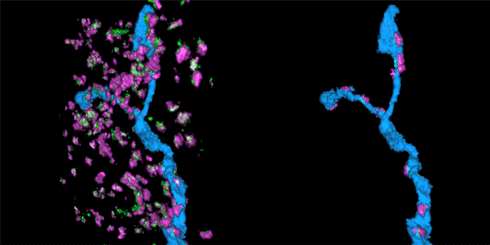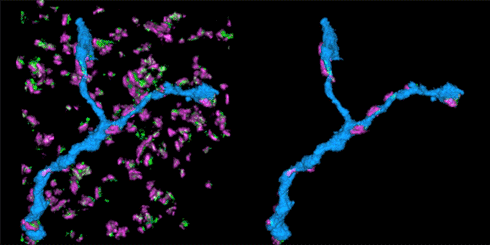
该STORM动画展示的是在抑制性突触中的树突分支。左边的动画展示的是在突触中的树突分支(蓝色),而右侧动画展示的是只有树突分支及与其连接的突触部分。
4.Chromosome Organization by a Nucleoid Associated Protein [2011, Science]

该STORM动画展示了一个正在分裂的细菌中的核糖体。核糖体用原位杂交的方法由光控染料所标记。研究人员利用超分辨率荧光显微镜结合染色体构象捕获分析法(chromosome-conformation capture assay)对活体大肠杆菌细胞内的拟核相关蛋白(nucleoid-associated proteins ,NAPs)进行了跟踪观察,并由此揭示了细菌遗传物质组织机制。
参考文献
[1]Rust, M.J., M. Bates, and X. Zhuang, Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Methods, 2006. 3(10): p. 793-5.
[2]Rasnik, I., S.A. McKinney, and T. Ha, Nonblinking and long-lasting single-molecule fluorescence imaging. Nat Methods, 2006. 3(11): p. 891-3.
[3]Betzig, E., et al., Imaging intracellular fluorescent proteins at nanometer resolution. Science, 2006. 313(5793): p. 1642-5.



